Technologie SNAP-tag
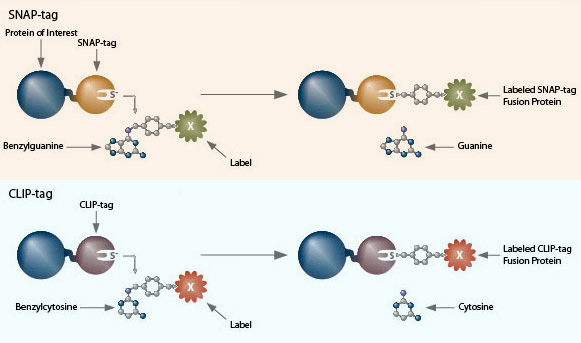
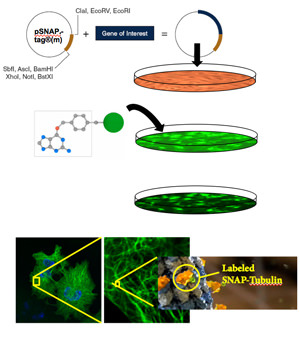
Les systèmes de marquage de protéines SNAP-tag et CLIP-tag permettent de lier à une protéine d’intérêt, de façon spécifique et covalente, n’importe quelle molécule ou presque. Cette méthode comporte deux étapes : clonage et expression de la protéine d’intérêt fusionnée avec un SNAP-tag®, puis marquage de la protéine de fusion avec le substrat du SNAP-tag de votre choix. Le SNAP-tag est une petite protéine fondée sur l’O6-alkylguanine-ADN-alkyltransférase (hAGT) humaine, une protéine de réparation de l’ADN.
Les substrats du SNAP-tag sont des colorants, des fluorochromes, la biotine ou des billes couplés aux groupes partants de la guanine ou de la chloropyrimidine via un lieur benzyle. Lors de la réaction de marquage, le groupe benzyle substitué venant du substrat se lie au SNAP-tag de manière covalente. Le CLIP-tag™ est une version modifiée du SNAP-tag conçue pour réagir avec la benzylcytosine plutôt que les dérivés de la benzylguanine. L’utilisation en parallèle du SNAP-tag et du CLIP-tag permet le marquage simultané, indépendant et complémentaire de deux protéines dans les mêmes cellules.
Applications
- Marquage simultané de deux protéines dans des cellules vivantes
- Études de localisation et de translocation des protéines
- Analyses pulse-chase
- Études d’internalisation des récepteurs
- Marquage sélectif de la surface cellulaire
- Analyses pull-down
- Détection de protéines en SDS-PAGE
- Cytométrie en flux
- Études de liaison à haut débit en microplaques
- Analyses d’interactions avec des biocapteurs
- Études de liaison par FRET
- Marquage d’une molécule unique
- Microscopie à super-résolution
SNAP-tag : un outil d’étiquetage polyvalent pour l’étude de la dynamique des protéines et bien plus encore
 Brochure:
Brochure:
Prix Nobel de chimie 2014
Le prix Nobel de chimie 2014 a été décerné à Stefan Hell (Göttingen, Allemagne), qui a utilisé le SNAP-tag dans ses études de microscopie STED :
- Stimulated emission depletion nanoscopy of living cells using SNAP-tag fusion proteins (Nanoscopie à déplétion par émission stimulée sur cellules vivantes à l’aide de protéines de fusion contenant le SNAP-tag) ;
- Multicolor fluorescence nanoscopy in fixed and living cells by exciting conventional fluorophores with a single wavelength (Nanoscopie à fluorescence multicolore sur cellules vivantes fixées, avec excitation de fluorochromes conventionnels par une longueur d’onde unique).

Cellules U2-OS en microscopie confocale vs STED :
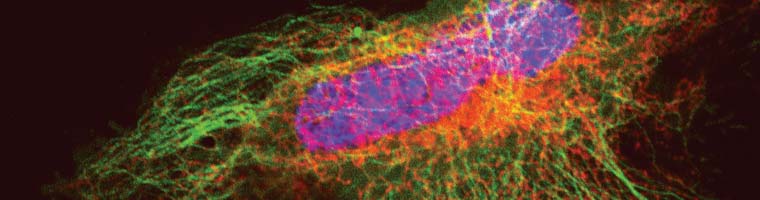
Les cellules surexpriment une protéine de fusion SNAP-Cep41 (une protéine qui se lie aux microtubules), détectée à l’aide de SNAP-Cell® 647SiR.
Lukinavičius G et al. (2013) « A near-infrared fluorophore for live-cell super-resolution microscopy of cellular proteins. » Nat. Chem.5(2) : 132-9.
Comparaison des technologies SNAP-tag®/CLIP-tagTM avec la GFP
Les technologies SNAP-tag/CLIP-tag sont complémentaires à la GFP, mais présentent des avantages par rapport à cette dernière dans plusieurs applications.
| Application | SNAP-tag/CLIP-tag | GFP et autres protéines fluorescentes |
| Time-resolved fluorescence | Fluorescence can be initiated upon addition of label | Color is genetically encoded and always expressed. Also, photoactivatable fluorescent proteins require high intensity laser light, which may activate undesired cellular pathways (e.g., apoptosis) |
| Pulse-chase analysis | Labeling of newly synthesized proteins can be turned off using available blocking reagents (e.g., SNAP-Cell® Block) | Fluorescence of newly synthesized proteins cannot be quenched to investigate dynamic processes |
| Ability to change colors | A single construct can be used with different dye substrates to label with multiple colors | Requires separate cloning and expression for each color |
| Surface specific labeling | Can specifically label subpopulation of target protein expressed on cell surface using non-cell permeable substrates | Surface subpopulation cannot be specifically visualized |
| Visualizing fixed cells | Resistant to fixation; strong labeling | Labile to fixation; weak labeling |
| Pull-down studies | “Bait” proteins can be covalently captured on BG beads | Requires anti-GFP antibody to non-covalently capture “bait” protein, complicating downstream analysis |
| Live animal imaging | Near-IR dyes are available, permitting deep tissue visualization | Limited to visible wavelengths |
Troubleshooting
Application |
Problem |
Possible Cause |
Solution |
Cellular Labeling |
No labeling | Fusion protein not expressed |
|
| Weak labeling | Poor expression and/or insufficient exposure of fusion protein to substrate |
|
|
| Rapid turnover of fusion protein |
|
||
| High background | Non-specific binding of substrates |
|
|
| Signal strongly reduced after short time | Instability of fusion protein |
|
|
| Photobleaching |
|
||
Labeling in Solution |
Precipitation | Insoluble fusion |
|
| Weak or no labeling | Exhaustive labeling has not been achieved |
|
|
| Loss of activity | Instability of fusion protein |
|
Starter Kits
| Product | NEB#. | Plasmid | Fluorophore | Block | Applications | Price |
| SNAP-Cell® Starter Kit | E9100S | pSNAPf Vector | SNAP-Cell® 505, SNAP-Cell® TMR-Star | SNAP-Cell® Block | – Intracellular labeling – Cell surface labeling – in vitro analysis | 252 € |
| SNAP-Surface® Starter Kit | E9120S | pSNAPf Vector | SNAP-Surface® 488, SNAP-Surface® 549 | SNAP-Surface™ Block | – Cell surface labeling – in vitro analysis | 252 € |
| CLIP-Cell™ Starter Kit | E9200S | pCLIPf Vector | CLIP-Cell™ 505, CLIP-Cell™ TMR-Star | CLIP-Cell™ Block | – Intracellular labeling – Cell surface labeling – in vitro analysis | 252 € |
| CLIP-Surface™ Starter Kit | E9230S | pCLIPf Vector | CLIP-Surface™ 488, CLIP-Surface™ 547 | CLIP-Cell™ Block | – Cell surface labeling – in vitro analysis | 252 € |
| ACP-Surface Starter Kit | E9300S | pACP-tag(m)-2 Vector | CoA 488, CoA 547 | N/A | – Cell surface labeling – in vitro analysis | 252 € |
Publications
STED
Guzmán, C. et al. (2014) « The efficacy of Raf kinase recruitment to the GTPase H-ras depends on H-ras membrane conformer specific nanoclustering » J. Biol. Chem. 289, 9519-9533.
Stagge, F. et al. (2013) « Snap-, CLIP- and Halo-Tag Labelling of Budding Yeast Cells » PLoS One 8(10): e78745.
Lukinavičius, G. et al. (2013) « Selective Chemical Crosslinking Reveals a Cep57-Cep63-Cep152 Centrosomal Complex » Curr. Biol. 23, 265-270.
Lukinavičius, G. et al. (2013) « A near-infrared fluorophore for live-cell super-resolution microscopy of cellular proteins » Nat. Chem. 5, 132-139.
Pellett P. A. et al. (2011) « Two-color STED microscopy in living cells. » Biomed. Opt. Expr. 2, 2364-2371.
Testa I. et al. (2010) « Multicolor Fluorescence Nanoscopy in Fixed and Living Cells by Exciting Conventional Fluorophores with a Single Wavelength » Biophys. J. 99, 2686-94.
Hein B. et al. (2010) « Stimulated Emission Depletion Nanoscopy of Living Cells Using SNAP-Tag Fusion Proteins. » Biophys. J. 98, 158–163.
STORM
Liu, Z. et al. (2014) « Super-resolution imaging and tracking of protein-protein interactions in sub-diffraction cellular space » Nat. Commun. 5, 4443.
Perkovic, M. et al. (2014) « Correlative Light- and Electron Microscopy with chemical tags » J. Struct. Biol. 186, 205-213.
Carlini, L. et al. (2014) « Reduced Dyes Enhance Single-Molecule Localization Density for Live Superresolution Imaging » ChemPhysChem 15, 750-755.
Sateriale, A. et al. (2013) « SNAP-Tag Technology Optimized for Use in Entamoeba histolytica » PLoS One 8(12), e83997.
Lukinavičius, G. et al. (2013) « A near-infrared fluorophore for live-cell super-resolution microscopy of cellular proteins » Nat. Chem. 5, 132-139.
Malkusch, S. et al. (2013) « Single-molecule coordinate-based analysis of the morphology of HIV-1 assembly sites with near-molecular spatial resolution » Histochem. Cell Biol. 139, 173-179.
van de Linde, S. et al. (2011) « Direct stochastic optical reconstruction microscopy with standard fluorescent probes » Nat. Protoc. 6, 991-1009.
Eckhardt M. et al. (2011) « A SNAP-Tagged Derivative of HIV-1-A Versatile Tool to Study Virus-Cell Interactions. » PLoS One 6(7), e22007.
Jones S. A. et al. (2011) « Fast, three-dimensional super-resolution imaging of live cells. » Nat. Methods 8, 499-505.
Klein T. et al. (2011) « Live-cell dSTORM with SNAP-tag fusion proteins. » Nat. Methods 8, 7-9.
Dellagiacoma C. et al. (2010) « Targeted Photoswitchable Probe for Nanoscopy of Biological Structures » ChemBioChem 11, 1361–1363.
PALM
Benke, A. et al. (2012) « Multicolor Single Molecule Tracking of Stochastically Active Synthetic Dyes » Nano Lett. 12, 2619-2624.
Banala, S. et al. (2012) « A caged, localizable rhodamine for superresolution microscopy » ACS Chem. Biol. 7, 289-293
RLS-SRM
Zhao, Z. W. et al. (2014) « Spatial organization of RNA polymerase II inside a mammalian cell nucleus revealed by reflected light-sheet superresolution microscopy » Proc. Natl. Acad. Sci. USA 111, 681-686.