Les kits NEBNext ARTIC répondent au besoin croissant de méthodes fiables, précises et rapides pour le séquençage du SARSCoV-2, avec Oxford Nanopore Technologies ou Illumina. Développés en collaboration avec le réseau ARTIC (artic. network). ces kits utilisent l’approche ARTIC pour le séquençage multiplexe par amplicon de génome viral complet. Les kits contiennent des pools d’amorces V3 équilibrés pour une couverture plus uniforme du génome et sont efficaces avec 10 à 10 000 copies de génome du SARS-CoV-2. Les réactifs et les protocoles pour la RT-PCR et la préparation de banques ont tous été spécialement optimisés pour les flux de travail SARS-CoV-2 ARTIC.
Nouveau Primer Set inclus pour une meilleure couverture des variants SARS-CoV-2 ainsi que de nouveaux protocoles Express
Depuis septembre 2021, 2 options de primers sont disponibles dans le NEBNext ARTIC SARS-CoV-2 FS Library Prep Kit (Illumina) (NEB #E7658), NEBNext ARTIC SARS-CoV-2 Companion Kit (Oxford Nanopore Technologies) (NEB #E7660) et NEBNext ARTIC SARS-CoV-2 RT-PCR Module (NEB #E7626) :
- Les pools de primers V3 ARTIC ont été balancés via la méthodologie développée par NEB bâtie à partir de données empiriques issues de séquençage. Combinés à des réactifs optimisés pour la RT-PCR, les kits offrent une uniformité de rendement accrue à partir de gRNA sur un large spectre de nombre de copies.
- Les VarSkip (pour Variant Skip) Short primers ont été dessinés par NEB afin de réduire l’impact des variants du SARS-CoV-2 sur l’amplification et en accroître la performance, y compris avec les variants Omicron et Delta. Le variant Omicron peut être séquencé grâce aux NEBNext VarSkip Short (VSS) primers. A noter : 2 dropouts (amplicons 56 et 67) et 2 amplicons (20 et 64) avec une plus faible couverture. Réaliser 2 libraries, avec ARTIC V3 et VSS, permet une couverture génomique quasi complète. Les pools de primers ont été équilibrés pour uniformiser la couverture.
Information sur les chevauchements de primer pour les variants Omicron et Delta
Les deux kits compatibles avec le séquençage Illumina génèrent des inserts de ~150 pb (pour un séquençage 2 x 75) ou ~400 pb (pour un séquençage 2 x 250), et comportent une formulation d’ADN polymérase innovante qui élimine la nécessité de normaliser les concentrations d’amplicons avant la préparation des banques.
- Couverture plus uniforme du génome du SARS-CoV-2
- Efficace avec un large éventail de quantités de génome viral initial (10–10 000 copies)
- Des conditions de RT uniques pour toutes les quantités d’échantillons initiaux
- Pas de normalisation des amplicons avant la préparation des banques (kits compatibles avec Illumina)
- NEBNext Sample Purification Beads (SPRIselect®) fournies
- Excellent rapport qualité/prix
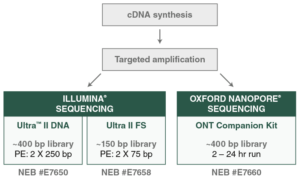
Options du NEBNext ARTIC Kit:
Flux de travail du NEBNext ARTIC Kit :
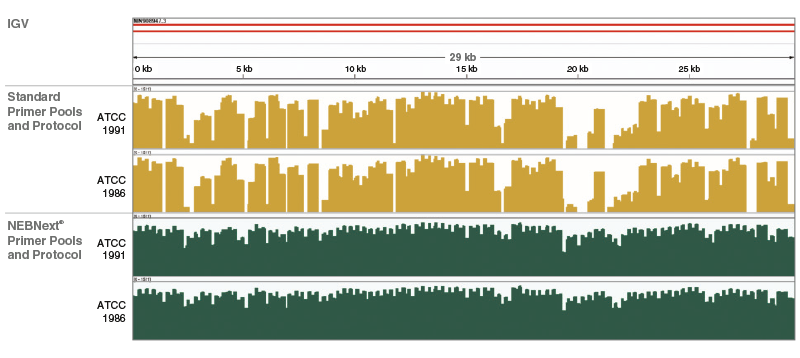
Couverture du génome avec NEBNext ARTIC Kit :
La visualisation de la couverture des lectures du génome du SARS-CoV-2 dans Integrative Genome Viewer montre une couverture supérieure et plus uniforme du génome avec les amorces, réactifs et protocoles NEBNext. Pour les données expérimentales détaillées, veuillez consulter www.neb.com/E7660
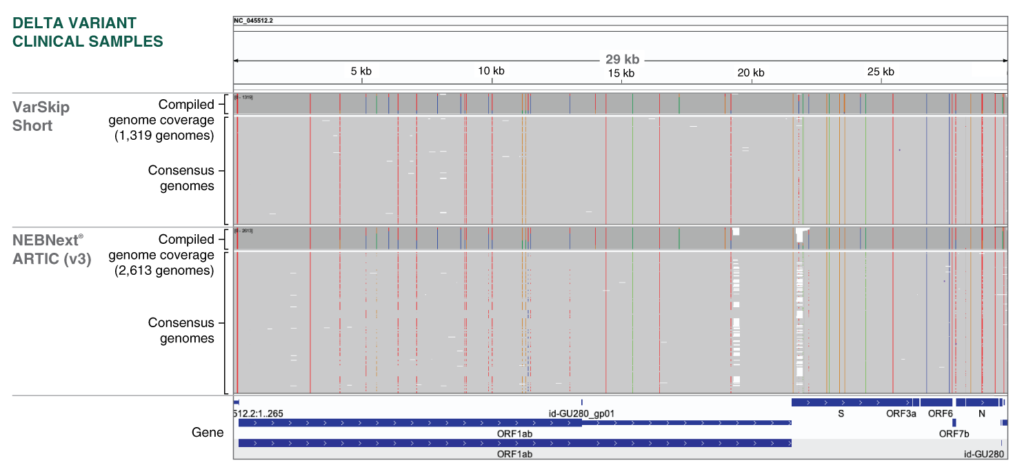
Amélioration de la couverture de séquence d’échantillons cliniques de SARS-CoV-2 Delta avec les VarSkip Short primers :
Séquences consensus classifiées comme B.1.617.2 (Delta) dans le Massachusetts (où les VarSkip Short ont été testés sur le terrain) collectées à partir de NBCBI Virus [1] et divisées en séquences VarSkip Short 2,613 ARTIC V3 et 1,319 via le champ SRA “design » (NEB_VarSkip_v1 = VarSkip Short, présumé par d’autres comme étant ARTIC V3). Les Reads ont été alignés avec la référence NC_045512.2 via minimap2 (minimap2 -x asm5) et visualisés avec IGV. Moins de dropouts observés pour les séquences consensus générées à partir d’amplicons VarSkip. Données expérimentales détaillées sur www.neb.com/E7658
|
„We have been impressed by the great performance of the NEBNext ARTIC FS kit in our sequencing of human samples containing SARS-CoV-2 RNA. With this kit we have been successfully processing samples of varying quality and quantity and have been amazed by its sensitivity and remarkable uniformity of the viral genome coverage. The quick turnaround time of the robust workflow has been truly beneficial for us.“ Dr. Vladimir Benes, Head of GeneCore EMBL Heidelberg, Early-Access-User |
||||
 |
||||
Liste des Produits
As of: 14.05.2021
Plus d’informations disponibles dans la section Ressources Techniques ou sur neb.com