Une alternative performante au séquençage de bisulfite pour l’analyse du méthylome
Le séquençage de génome entier après traitement au bisulfite (WGBS, Whole Genome Bisulfite Sequencing) constitue depuis longtemps la technique de référence pour l’analyse du méthylome, mais la réaction chimique avec le bisulfite endommage et dégrade l’ADN, entraînant sa fragmentation et sa perte. En outre, les banques ainsi obtenues présentent un biais GC important et sont enrichies en régions méthylées.
Afin de remédier à ces problèmes, nous avons développé une nouvelle approche enzymatique, NEBNext Enzymatic Methyl-seq (EM-seq), pour l’identification de la 5-méthylcytosine (5-mC) et de la 5-hydroxyméthylcytosine (5-hmC).
- Sensibilité élevée pour la détection des 5-mC et des 5-hmC
- Excellente efficacité d’alignement
- Couverture uniforme des GC
- Détection d’un maximum de CpG avec un minimum de lectures de séquençage
- Distribution uniforme des dinucléotides
- Tailles d’inserts accrues
- Préparation efficace de banques
- Module de conversion disponible séparément
Cette méthode permet de minimiser l’endommagement de l’ADN grâce à une conversion enzymatique hautement efficace et de produire des librairies de qualité avec les réactifs de préparation de banques NEBNext UltraTM II fournis, pour une détection optimale des 5-mC et des 5-hmC à partir d’un minimum de lectures de séquençage. Pratique : avec la méthode EM-seq, vous obtenez la même séquence convertie qu’avec le WGBS et pouvez ainsi utiliser les mêmes techniques d’analyse.
Video: EM-seq Workflow
Envie d’essayer le nouveau NEBNext Enzymatic Methyl-seq ? Demandez un échantillon dès aujourd’hui :
Avis de nos premiers utilisateurs :
|
„Em-Seq (…) enables us to determine in a precise and DNA sparing way the cytosine methylation status even at low integrity DNA. (…) It also opens new avenues to explorations of methylation at intact long DNA fragments.“ Vladimir Benes, Head Genomics Core Facility at EMBL Heidelberg |
||||
 |
||||
|
„Enzymatic conversion of EM-Seq is THE alternative and our comparisons clearly showed, that the quality of data obtained is better than with conventional bisulfite conversion.“ Dr. Alexander Vogt, Sequencing Specialist, Vienna BioCenterCore Facilities |
||||
 |
||||
Une nouvelle alternative au séquençage au bisulfite
La méthode NEBNext EM-seq peut être utilisée pour différentes plateformes de séquençage :
Plateformes Illumina :
NEBNext Enzymatic Methyl-seq Kit (réf. E7120) contient tous les réactifs nécessaires à la conversion enzymatique de l’ADN méthylé et à la préparation de banques pour le séquençage Illumina.
Autres plateformes :
NEBNext Enzymatic Methyl-seq Conversion Module (réf. E7125) peut être utilisé pour la conversion enzymatique de l’ADN en vue de la préparation de banques.
Flux de travail NEBNext Enzymatic Methyl-seq Kit (réf. E7120) pour Illumina :
Les banques sont préparées à partir de seulement 10 ng d’ADN initial, des réactifs NEBNext Ultra II fournis et de l’EM-seq Adaptor optimisé. TET2 oxyde ensuite les 5-mC et les 5-hmC, les protégeant ainsi de la désamination par APOBEC à l’étape suivante. En revanche, les cytosines non modifiées sont désaminées en uraciles. Les banques sont amplifiées à l’aide d’un master mix NEBNext de Q5U (une version modifiée de Q5 High-Fidelity DNA Polymerase), puis séquencées sur un instrument Illumina.
Offrant une conversion robuste et une réduction de l’endommagement de l’ADN, le protocole EM-seq se combine avec la méthode hautement efficace de préparation de banques NEBNext Ultra II, pour une excellente détection des CpG à partir d’un minimum de lectures de séquençage.
Le multiplexage en toute simplicité :
NEBNext Enzymatic Methyl-seq Kit (réf. E7120) contient des couples d’amorces double index uniques, les EM-seq Index Primers, pour le multiplexage de 24 échantillons. Les amorces 96-well NEBNext Multiplex Oligos for Enzymatic Methyl-seq (Unique Dual Index Primer Pairs) (réf. E7140L) peuvent être utilisées en supplément pour amplifier jusqu’à 120 échantillons en multiplexe.
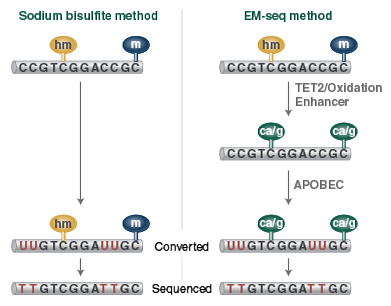
Le traitement de l’ADN au bisulfite de sodium entraîne la désamination des cytosines en uraciles, tandis que les cytosines modifiées (5-mC et 5-hmC) ne sont pas désaminées. Lorsque l’ADN traité au bisulfite est amplifié par PCR, les uraciles sont remplacés par des thymines, et les 5-mC et 5-hmC par des cytosines. Après séquençage, les cytosines non méthylées sont représentées par des thymines, et les 5-mC et 5-hmC par des cytosines. La comparaison des séquences avec des séquences de référence (génome ayant subi des conversions C/T et G/A) permet de déterminer l’état de méthylation.
Durant la première étape de conversion de la méthode EM-seq, TET2 et l’Oxidation Enhancer (un agent favorisant l’oxydation) sont utilisés pour protéger les cytosines modifiées contre la désamination en aval. TET2 assure l’oxydation enzymatique des 5-mC et 5-hmC en 5-carboxycytosines (5-caC) via une réaction en cascade. Les 5-mC et les 5-hmC sont ainsi protégées de la désamination. Les 5-hmC peuvent également être protégées de la désamination par glucosylation en présence de l’Oxidation Enhancer pour former des 5-glucosyl-hydroxyméthylcytosines (5-ghmC). Durant la seconde étape enzymatique, APOBEC est utilisée pour désaminer les cytosines sans affecter les 5-caC et les 5-ghmC. La séquence convertie qui en résulte est identique à celle de l’ADN traité au bisulfite et peut donc être analysée de la même manière.
Avantages de la méthode NEBNext Enzymatic Methyl-seq
Des inserts plus longs
Le flux de travail EM-seq comporte des étapes de traitement plus délicates que la conversion au bisulfite, pour un endommagement minimal de l’ADN. L’ADN converti par la méthode EM-seq est donc plus intact que celui traité au bisulfite, ce qui permet d’obtenir des banques avec un taux supérieur d’inserts plus longs, comme illustré ci-dessous. Résultats : des lectures de séquençage plus longues, des alignements plus fiables et potentiellement des coûts de séquençage par base réduits, en fonction de l’instrument utilisé et d’autres paramètres de séquençage.
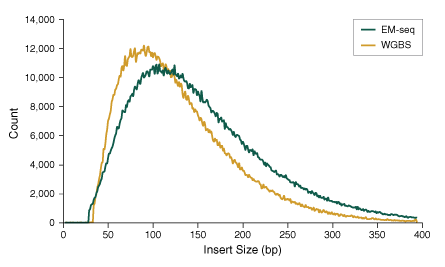
50 ng d’ADN génomique humain NA12878 ont été fragmentés en séquences de 300 pb à l’aide de l’instrument Covaris® S2 et utilisés comme échantillon initial dans les protocoles EM-seq et WGBS. Pour le WGBS, les banques ont été construites avec les réactifs NEBNext Ultra II, puis Zymo Research EZ DNA Methylation-GoldTM Kit a été utilisé pour la conversion au bisulfite. Les librairies ont été séquencées sur Illumina® MiSeq® (2 x 76 bases) et les tailles des inserts ont été déterminées à l’aide de Picard 2.18.14. La représentation graphique de la fréquence normalisée de chaque taille d’inserts montre que ces derniers sont plus longs avec la méthode EM-seq qu’avec le WGBS, ce qui indique que le protocole EM-seq n’endommage pas autant l’ADN que le traitement au bisulfite du WGBS.
Des rendements supérieurs
L’endommagement, la fragmentation et la perte d’ADN liés au traitement par le bisulfite réduisent les rendements après amplification des librairies converties au bisulfite. En revanche, le traitement plus doux lors de la conversion enzymatique EM-seq permet d’obtenir des banques d’ADN de qualité pouvant être amplifiées de manière efficace. Ainsi, les rendements obtenus avec la méthode EM-seq sont plus élevés que ceux des banques WGBS, avec un nombre réduit de cycles PCR.
En outre, ces rendements supérieurs ne sont pas dus à la présence de doublons de PCR, particulièrement évidente aux faibles quantités d’échantillons initiaux. En effet, les banques EM-seq présentent des taux de doublons systématiquement plus faibles sur un large éventail de quantités d’échantillons initiaux.
Une couverture uniforme des GC
La réussite d’un séquençage dépend non seulement du rendement, mais aussi de la qualité de la banque. Une librairie de qualité offre une représentation uniforme de l’échantillon initial, avec une couverture homogène quel que soit le taux de GC.
Or, la conversion au bisulfite, un traitement agressif qui agit sur les cytosines non méthylées – c’est-à-dire la majorité des cytosines – et les endommage, affecte les régions riches en GC de manière inégale. Avec cet endommagement, ce fractionnement et cette perte biaisés, les banques traitées au bisulfite sont donc sous-représentées en GC et sur-représentées en AT. En revanche, les banques EM-seq présentent une couverture uniforme des GC, ce qui montre que l’ADN n’est pas endommagé et que ces librairies sont représentatives de l’échantillon initial.
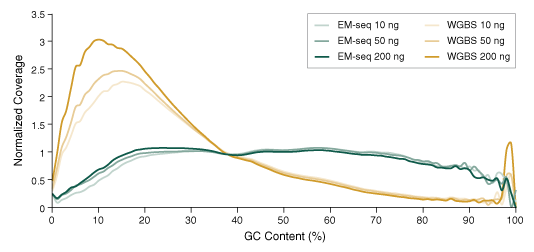
10, 50 et 200 ng d’ADN génomique humain NA12878 ont été fragmentés en séquences de 300 pb à l’aide de l’instrument Covaris S2 et utilisés comme échantillons initiaux dans les protocoles EM-seq et WGBS. Pour le WGBS, les banques ont été construites avec les réactifs NEBNext Ultra II, puis Zymo Research EZ DNA Methylation-Gold Kit a été utilisé pour la conversion au bisulfite. Les librairies ont été séquencées sur Illumina NovaSeq® 6000 (2 x 100 bases). Les lectures ont été alignées sur hg38 à l’aide de bwa-meth 0.2.2. La couverture des GC a été analysée à l’aide de Picard 2.18.14 et la distribution de la couverture normalisée pour les différents taux de GC du génome (0-100 %) a été représentée graphiquement. Les banques EM-seq présentent une couverture des GC bien plus uniforme et ne sont ni sur-représentées en AT ni sous-représentées en GC, contrairement aux librairies WGBS.
Pour plus d’informations sur les performances, consulter ou télécharger notre note technique.
Les produits NEBNext Enzymatic Methyl-seq
As of: 07.05.2019
Les produits NEBNext sont disponibles à des tarifs compétitifs ne pouvant pas faire l’objet de remises supplémentaires.
Plus d’informations disponibles dans la section Ressources Techniques ou sur neb.com


