OneTaq DNA Polymerase est un mélange optimisé de Taq et de Deep Vent DNA Polymerase, conçu à la fois pour les PCR de routine et les amplifications difficiles. L’activité exonucléase 3′→5′ de la Deep Vent DNA Polymerase améliore la fidélité et la robustesse de la Taq DNA Polymerase. OneTaq Standard Reaction Buffer, OneTaq GC Reaction Buffer et High GC Enhancer ont été formulés pour offrir de bons rendements avec une optimisation minimale, quel que soit le taux de GC de la matrice.
OneTaq DNA Polymerase est livrée avec deux tampons 5X (Standard et GC) et une solution High GC Enhancer. Pour la plupart des amplicons classiques et/ou riches en AT (ADN du phage lambda, etc.) ou les séquences complexes composées jusqu’à ~65 % de GC, OneTaq Standard Reaction Buffer permet d’obtenir une amplification robuste. Pour les matrices riches en GC, OneTaq GC Reaction Buffer peut améliorer aussi bien les performances que le rendement. Pour les séquences avec des taux de GC particulièrement élevés ou difficiles à amplifier, OneTaq High GC Enhancer peut être ajouté à une concentration finale de 10-20 % aux réactions contenant OneTaq GC Reaction Buffer.
Nouveau :
OneTaq Quick-Load DNA Polymerase – Chargement direct sur gel après la PCR !
Outre le tampon réactionnel classique, cette nouvelle version contient un Quick-Load Reaction Buffer coloré, pour un chargement direct sur gel d’agarose après la PCR. OneTaq Quick-Load DNA Polymerase est donc idéale pour les applications PCR classiques telles que le génotypage, la PCR sur colonie, etc. (pour des amplicons allant jusqu’à 6 kb et environ 55 % de GC).
Rapide, fiable et avantageux !
Quelle que soit l’option que vous choisissez, OneTaq DNA Polymerase est l’enzyme de référence pour vos PCR au quotidien.
Avantage :
- Idéale pour les applications PCR classiques et sur matrices riches en AT ou GC
Applications :
- PCR à haute sensibilité
- PCR à haut débit
- PCR de routine
- PCR sur matrices riches en GC
- PCR sur matrices riches en AT
- PCR sur colonie
- PCR longue (jusqu’à ~6 kb d’ADN génomique)

Note d’Application :
« Robust Colony PCR from Multiple E. coli Strains using OneTaq® Quick-Load® Master Mixes »
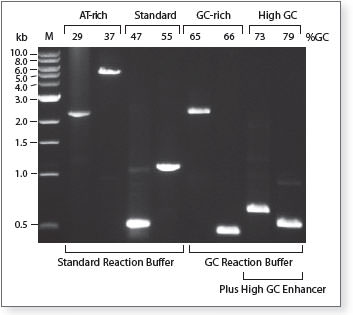
Amplification de différentes séquences d’ADN génomique humain et de C. elegans avec différents taux de GC à l’aide de OneTaq DNA Polymerase. Le pourcentage de GC est indiqué au-dessus du gel. La piste M correspond au 1 kb DNA Ladder
OneTaq Hot-Start DNA Polymerase
OneTaq® Hot Start DNA Polymerase est un mélange optimisé de Taq DNA Polymerase, de Deep Vent® DNA Polymerase et d’un aptamère inhibiteur.
Ce dernier se lie de manière réversible à la polymérase et inhibe son activité à des températures inférieures à 45 °C. L’enzyme est activée dans les conditions normales de thermocyclage, ce qui permet de préparer les réactions à température ambiante. OneTaq Hot Start DNA Polymerase ne nécessite pas d’étape d’activation thermique séparée et peut être utilisée dans les protocoles PCR habituels utilisant la Taq ADN polymérase.
Des périodes prolongées d’incubation à température ambiante n’ont aucune influence sur les performances de OneTaq® Hot Start DNA Polymerase
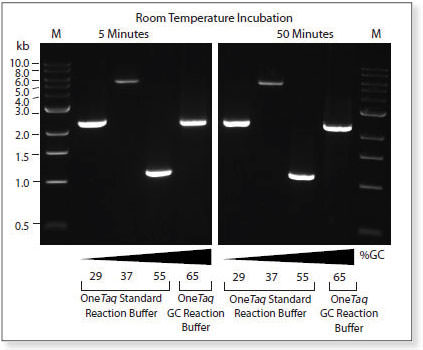
Amplification de différentes séquences d’ADN génomique humain et de C. elegans avec différents taux de GC à l’aide de OneTaq Hot Start DNA Polymerase. La présence ou l’absence d’une incubation prolongée à température ambiante n’affecte pas les performances. Le pourcentage de GC est indiqué en dessous du gel. La piste M correspond au 1 kb DNA Ladder.
Comparison of recommended activation steps of commercially available Hot Start DNA Polymerases
| MANUFACTURER | ENZYME | ACTIVATION STEP* | HOT START FORM |
| Applied BioSystems | AmpliTaq Gold® 360 | 10′, 95°C | Modified |
| Invitrogen | Platinum® Taq | 30″–2′, 94°C | Ab |
| Promega | GoTaq® Hot Start | 2′, 94–95°C | Ab |
| Qiagen | HotStarTaq | 15′, 95°C | Modified |
| Roche | FastStart Taq | 4′, 95°C | Modified |
| Sigma | JumpStart™ Taq | 1′, 94°C | Ab |
| Thermo Fisher | Thermo-Start Taq | 15′, 95°C | Modified |
| NEB | OneTaq | None | Aptamer |
Liste de produits
Plus d’informations disponibles dans la section Ressources Techniques ou sur neb.com